Sujet de thèse
Titre: Analyse automatique d’images de réseau de microtubules pour la prédiction d’interaction de protéines
Mot-clés : Apprentissage automatique, Réseau de neurones profond, Segmentation d’image, Application à la bioinformatique.
Société associée: Synsight
Contexte
Les interactions entre protéines dans les cellules humaines sont multiples et complexes. Pourtant, mieux
comprendre ces interactions est nécessaire pour découvrir de nouveaux médicaments, en particulier lorsque ceuxci
ciblent spécifiquement des protéines. Les développements technologiques visant à mieux comprendre les
interactions entre protéines sont donc d’un intérêt majeur pour développer de nouvelles stratégies thérapeutiques.
Un objectif du laboratoire SABNP (Inserm U1204) est de développer des technologies de rupture d’études des
interactions entre molécules biologiques (protéines, ARN, ADN,…). Dans ce cadre, le laboratoire SABNP a élaboré
une nouvelle méthode grâce à laquelle les interactions entre protéines peuvent être observées et quantifiées,
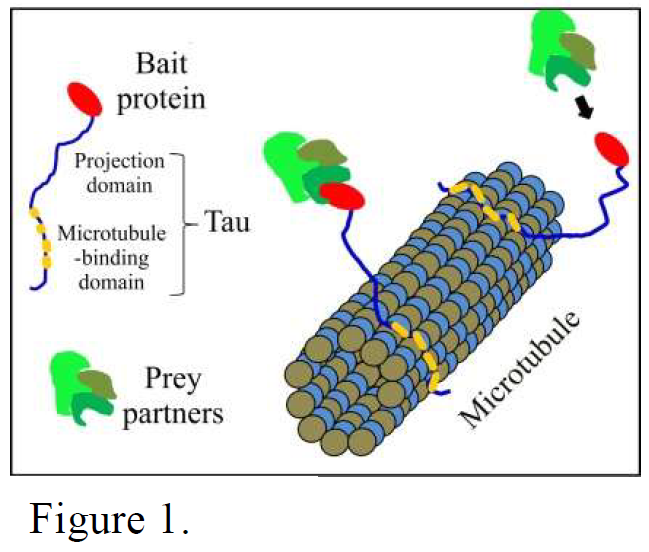
directement au sein de la cellule. La détection est basée sur la présence de microtubules, de longs cylindres de rayon
nanométrique dont la morphologie caractéristique est facilement observable par microscopie à fluorescence. Ils
constituent une sorte de squelette de la cellule et forme un réseau de filament s’étendant à partir du centrosome.
Avec cette technologie (Microtubules Assay), le réseau de microtubules sert de plate-forme d’étude des interactions
entre protéines dans des cellules vivantes. Le principe est simple : une protéine servant d’« appât » est liée aux
microtubules par l’intermédiaire d’un « linker ». Lorsqu’une protéine « proie » interagit avec la protéine « appât »,
la présence de la protéine « proie » est détectée par fluorescence sur les microtubules (figure 1). Les chercheurs ont
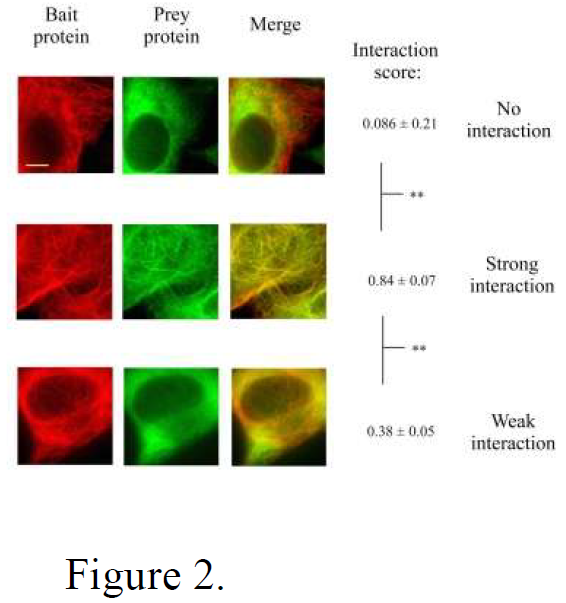
validé le potentiel de cette nouvelle méthode sur des cas d’interactions connues entre des protéines se liant à l’ARN
ou des protéines de la membrane cellulaire. Les interactions ont été clairement visualisées sur les microtubules de
cellules génétiquement modifiées exprimant l’ «appât» et la «proie» (Figure 2).
La société SYNSIGHT associée à ce projet a pour objectif d’améliorer les performances de la technologie du SABNP
en développant la capacité de criblage à très haut-débit axée et en utilisant des algorithmes d’apprentissage en vue
de découvrir de nouvelles molécules thérapeutiques et d’identifier de nouvelles applications de molécules existantes
par repositionnement sur cibles biologiques. La technologie du SABNP est tout à fait adaptée à des expériences de
High-content screening qui demandent une vitesse de détection très rapide, et est possible en cellules vivantes. Le
laboratoire IBISC, spécialisé en autre en analyse d’images et apprentissage automatique, collabore à ce projet pour
le développement la partie intelligence artificiel.
Les applications de cette technologie concernent la recherche médicale et le développement de nouveaux
médicaments. En effet, les interactions entre protéines jouant un rôle clé dans le fonctionnement des organismes
vivants, cette méthode promet de nombreuses applications, en particulier pour tester leur action ou découvrir de
nouveaux médicaments et pour mieux connaître les dérèglements associés aux maladies. Plus précisément :
– Elle offre la possibilité de cribler dans des cellules humaines l’efficacité de candidats médicaments agissant sur les
interactions entre protéines.
– Elle permet aussi d’explorer dans des cellules l’impact de mutations responsables de maladies, afin d’en
comprendre le mécanisme, puis de rechercher des molécules thérapeutiques capables de corriger l’effet de ces
mutations sur les interactions entre protéines.
Sujet de recherche
L’objectif principal de ce sujet de thèse est de proposer des méthodes pour analyser automatiquement les images
des microtubules et prédire les interactions entre protéines. Le travail consistera à réaliser les tâches suivantes :
– Segmentation des cellules à partir d’images comprenant plusieurs milliers de cellules.
– Segmentation des microtubules à partir d’images de la protéine appât qui peuvent être de qualité variable
(discontinuité, faible rapport signal/bruit) sur des cellules uniques
– Mesurer la colocalisation entre les deux « couleurs « représentant » les deux protéines à l’échelle de la
cellule unique
– Prédire les niveaux d’expression des protéines « appât » et « proie » à l’échelle de la cellule unique
– Prédire à haut débit des scores d’interaction et des paramètres biophysiques traduisant l’interaction
– A partir de données à haut débit généré par Synsight sur des systèmes connus, développer un pipeline
partant d’une image de plusieurs milliers de cellules vers la prédiction d’un score d’interaction des protéines
– Tester sur des systèmes réels et sur de milliers de composés ciblant des interactions connues.
L’étudiant devra commencer par faire un état de l’art précis des méthodes d’apprentissage profond liées aux
différentes tâches qu’il devra traiter. Pour chacune il devra sélectionner les méthodes les plus pertinentes et les
adaptes aux problèmes de la thèse. Une attention particulière sera portée aux méthodes convolutionnelles, qui ont
démontré leur potentiel en classification d’images. Les modèles seront construits et testés à partir des données
internes à Synsight. La solution proposée devra avoir à la fois une grande précision de prédiction et être robuste aux
différentes conditions d’acquisition des images. Il est important de noter que le travail demandé ne consiste pas
simplement à reprendre des modèles d’apprentissage profond afin de les réutiliser sur les données de l’entreprise.
Il sera demandé à l’étudiant de modifier en profondeur les algorithmes d’apprentissage afin de les adapter aux
images de cellules et de microtubules et d’intégrer les connaissances a priori du domaine.
Compétences requises
BAC+5 (validé ou en cours) en informatique ou mathématiques appliquées (Master 2 Recherche ou École
d’ingénieurs). Des bases solides en programmation (dans l’idéal python) et de bonnes connaissances en
apprentissage automatique sont requises. Une expérience en apprentissage profond et en analyse d’image serait
appréciée. Une bonne maîtrise de l’anglais est indispensable.
Modalités de candidature
Candidature sous forme de fichier pdf regroupant les informations suivantes :
CV
Lettre de motivation ciblée
Coordonnées de référents à contacter (joindre éventuellement lettres de recommandation)
Relevés de notes des deux dernières années d’étude
Enseignements suivis et validés durant les deux dernières années d’étude
- Date limite de candidature: 6 mai 2019
- Contacts:
- David Pastre (PR Univ. Evry, laboratoire SABNP), courriel: david.pastreATuniv-evry.fr
- Blaise Hanczar (PR Univ. Evry, laboratoire IBISC, équipe AROBAS), courriel: blaise.hanczarATuniv-evry.fr
- Vincent Vigneron (MCF HDR Univ. Evry, laboratoire IBISC, équipe SIAM), courriel: vincent.vigneronATuniv-evry.fr
- Société partenaire: SYNSIGHT
- Proposition de thèse au format PDF