Sujet : « Apprentissage profond appliqué aux données transcriptomiques : self-training, self-supervised, transfer learning »
Contexte
L’apprentissage profond (Deep Learning) est une avancée majeure de l’intelligence artificielle de ces dernières années. Cette approche de l’apprentissage automatique consiste à apprendre à un réseau de neurones de grande taille à réaliser une tâche de prédiction à l’aide d’un ensemble de données d’apprentissage. L’apprentissage profond s’est rapidement imposé comme un standard dans de plusieurs domaines en pulvérisant les records des précédentes méthodes de l’état de l’art. Ses domaines de prédilection sont principalement l’analyse d’images et le traitement du langage naturel. Un des futurs enjeux majeurs de cette approche est son application à la santé.
Nos thèmes de recherche se concentrent plus spécifiquement sur la prédiction de phénotypes (diagnostiques, pronostiques, réponse aux traitements,… ) à partir de données d’expression de gènes. Un verrou scientifique majeur à lever pour avancer dans ce domaine est l’apprentissage de réseaux de neurones à partir de jeux d’apprentissage de petite taille. Nous proposons deux stages liés à ce sujet à partir d’approche de self-training, self-supervised, transfer learning.
Sujet
L’analyse de données transcriptomiques par apprentissage profond est un domaine de recherche très récent. La grande majorité des articles publiés ont moins de deux ans et parmi eux seulement une poignée s’intéresse à la prédiction de phénotypes. La raison de ce faible nombre de travaux publiés actuellement provient du manque de grands jeux de données transcriptomiques disponibles dû à leur coût élevé d’acquisition. Alors que les réseaux de neurones profonds traitant des images ou du langage naturel sont construits à partir de plusieurs centaines de milliers ou millions d’exemples, les jeux de données transcriptomiques publiques contiennent très peu de patients (quelques milliers au mieux). À cause de ce faible nombre d’exemples, l’apprentissage des réseaux de neurones profonds se heurte à des problèmes de sur-apprentissage, le réseau apprend par coeur les données mais pas le concept sous-jacent.
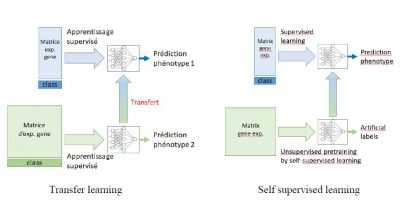
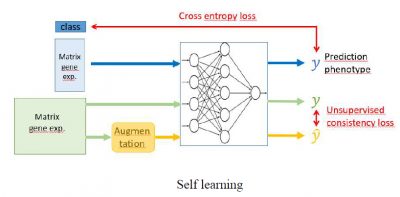
Dans ce sujet nous comptons pallier le problème de la petite taille des données d’apprentissage en utilisant différentes approches : self-training, self-supervised, transfer learning. Dans chaque cas, ces approches utilisent un jeu de données secondaire de grande taille (étiqueté ou non étiqueté). L’objectif est d’utiliser ces données secondaire afin d’apprendre une représentation optimale des données dans les couche caché du réseau et qui sera utilisée afin de rendre la tache de prédiction initiale plus facile.
Dans ce stage nous utiliseront ces méthodes pour transférer de l’information à travers plusieurs réseaux appris à partir de petit jeux de données transcriptomiques dans le but d’améliorer les performances de prédictions. Le travail consistera à faire un état de l’art et à sélectionner les méthodes d’apprentissage les plus performantes actuellement sur les données images. Puis il faudra adapter les approches sélectionnées pour une utilisation sur les données transcriptomiques. La dernière étape sera de tester les méthodes développées à travers une série d’expérimentations sur des jeux de données publiques.
Profils recherchés
Étudiant Master 2 Recherche ou en dernière année d’école d’ingénieur de formation informatique ou mathématiques appliquées.
Une solide formation en machine learning est indispensable.
Des bases en programmation python et une bonne maîtrise de l’anglais sont nécessaires.
Des connaissances en deep learning et programmation tensorflow / pytorch seraient appréciées.
Autonomie et curiosité pour la recherche scientifique.
Début du stage : 2021
Durée : 5-6 mois
Encadrant : Pr Hanczar Blaise
Pour postuler envoyer CV et relevé de notes à blaiseDOThanczarATibiscDOTuniv-evryDOTfr
- Date de l’appel : 12/11/2020
- Statut de l’appel : non pourvu
- Contact coté IBISC : Blaise HANCZAR (PR Univ. Évry, IBISC équipe AROBAS)
- Sujet de stage niveau Master 2 (format PDF)
- Web équipe AROBAS